In Zeiten von Corona ist das Projekt Folding@home ein wenig bekannter geworden. Wird doch mit diesem Projekt der Wissenschaft unter anderem bei der Erforschung von Corona unter die Arme gegriffen.
Folding@home
Folding@home ist eine Projekt, bei dem Computerbesitzer Forschungsgruppen helfen können, Proteinstrukturen von Krankheitsverursachern und deren Wechselwirkung mit anderen Proteinstrukturen zu entschlüsseln. Dabei handelt es sich nicht um eine klassische Proteinfaltung – also Synthetisierung der Aminosäureketten und anschließende Ausbildung der 3D-Struktur – sondern um eine Simulation, wie die schon gefalteten verschiedenen Strukturen auf andere Strukturen reagieren. Ich kann gerne einen Beitrag zum Thema Proteinsynthese und Proteinfaltung schreiben, wenn gewünscht und „das Internet“ als Quelle nicht ausreichen sollte.
Welche Krankheiten werden erforscht?
Ursprünglich wurden „nur“ Proteinstrukturen der Krankheiten Alzheimer, Krebs, Chorea Huntington und Parkinson simuliert. Aber inzwischen wird auch die Forschung über Struktur und die Wechselwirkungen von Corona unterstützt. Durch die aktuelle Corona-Pandemie bekam das Projekt daher riesigen Zulauf.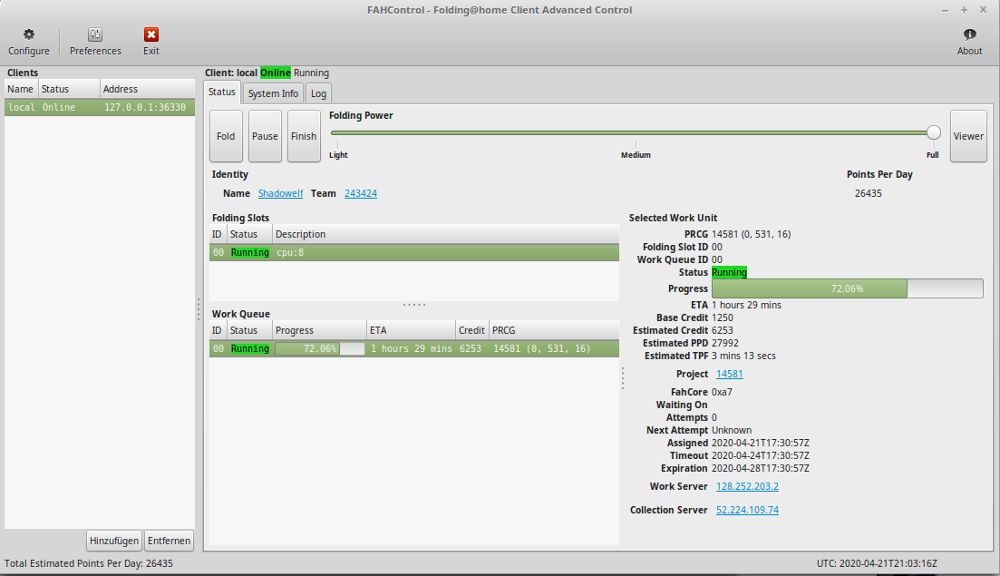
Folding@Home – Wie mache ich mit?
Ihr geht auf die Folding@Home-Download-Seite und bekommt automatisch den richtigen Client Download für euer Betriebssystem vorgeschlagen. Bei der einfachen Installation wird alles automatisch konfiguriert und die installierende Person muss höchstens sich eine Identität zulegen, damit sie auch wiedererkannt wird.
Arbeiten im Team bei Folding@Home
Bei Folding@Home gibt es die Möglichkeit einem Team beizutreten. Also wenn ihr mögt, gebt doch die Nummer 243424 an. Das ist die Nummer meines Teams des „Kuchen-Gank-Squad“. Wir sind schon fast auf Platz 8000 von über 250.000 Teams vorgedrungen – also beteiligt euch gerne. Hier der Direktlink zur Teamseite: Statistiken vom Kuchen Gank Squad.
Es sind schon einige Twitterer vertreten: @KatsuKatsumikat, @abraxa1982, @rubberduckweb, @_badmaxx_, @trancefish1 und @1Qursu. Sowie auch mehrere Personen über Instagram: @cljan69 und @otsihpem
Aber hier mal eine kleine Statistik der Top Five – leider kann ich noch nicht konfigurieren, wie viele Benutzer angezeigt werden. Da die Statistik-Server ab und zu Probleme haben, hoffe ich, dass das Plugin die Daten erfolgreich zieht:
[phoenix_folding_stats type=“team“ class=“table table-striped“ show_id=“yes“ show_donor_teams=“yes“ id=“243424″ show_logo=“no“ show_tagline=“no“]
Tipps & Tricks
Natürlich gibt es auch einige Tipps und Tricks, die ich nach und nach ergänzen werde:
- Corona wird miterforscht, in dem ihr in der Konfiguration im Reiter Advanced die „Cause Preferency“ auf „Any“ stehen lasst
- Wenn euch mal keine Einheit zugeteilt werden sollte und der nächste Server-Kontakt-Versuch sich schon im zweistelligen Minuten-Bereich befinden sollte: Rechtsklick in der Status-Übersicht auf das „Running“ und wählt „On Idle“ aus. Wartet ein paar Sekunden und Deselektiert das „On Idle“ wieder.
SETI@Home
Ein ähnliches Projekt SETI@Home gab es schon seit Ende des letzten Jahrhunderts. Mit diversen Clients – unter anderem einem Bildschirmschoner – wurden Datenpakete ausgewertet, um aus Radiosignalen aus dem Weltraum Spuren außerirdischer Kommunikation zu finden.
Beowulf-Cluster
Ein Beowulf-Cluster ist kein Online-Projekt. Aber hat mit der hiesigen Problematik insofern zu tun, als dass auch hier mehrere Rechner zusammen arbeiten. Eine Arbeitsgruppe am Oak Ridge National Laboratory benötigte 1996 kostbare Rechnerkapazitäten. Da gute Computer damals noch teurer waren als heute, behalfen sich die Forscher, in dem sie viele veraltete und deswegen nicht mehr benötigte Computer in einem Netzwerk zusammen schalteten. Hier ein Link zu einem Spektrum-Artikel zu diesem Thema.
Faltet ihr schon?
Faltet ihr schon Proteine mit Folding@Home? Beteiligt ihr euch bzw. eure Rechner an anderen Projekten mit verteilter Rechenkapazität? Oder an was für anderen interessanten Möglichkeiten beteiligt ihr euch?
Ein Gedanke zu „Folding@home – faltet ihr schon?“